13/06/2025 11h17
Macrófagos reciclam bactérias fagocitadas para abastecer seu próprio metabolismo
Texto produzido por Larissa Andrade
Seminário apresentado junto ao Centro de Pesquisa em Doenças Inflamatórias (CRID, Center for Research in Inflammatory Diseases).
Trabalho intitulado “Macrophages recycle phagocytosed bacteria to fuel immunometabolic responses” (1).
Células eucarióticas, de maneira geral, possuem preferência pela captação de moléculas de baixo peso molecular para atividades metabólicas (2). Algumas populações, entretanto, podem ativar vias alternativas para essa modulação, tal como é feito por macrófagos (2-4). Ainda, estas células modulam sua própria ativação ao identificar a viabilidade dos microrganismos por elas fagocitadas (5-7). O trabalho de Lesbats e colaboradores (2025) trouxe como problemática central qual seria o destino metabólico de bactérias depois de serem fagocitadas por macrófagos e a relevância deste processo para essa população celular.
O grupo inicialmente observou que, diante de estímulo com LPS ou killed E. coli (KEC), houve diferença em relação à genes diferencialmente expressos associados ao transporte de solutos, sendo que somente a KEC favoreceu a atividade de cadeia respiratória mitocondrial, obtido por meio da redução de taxa de consumo de oxigênio (OCR). Por meio da utilização de carbono associado a isótopo marcados, observou-se nos macrófagos a presença de moléculas provenientes das bactérias fagocitadas, especialmente na via de síntese de glutationa.
Por já ser descrito na literatura o envolvimento do complexo mTORC1 na reutilização de moléculas obtidas pela fagocitose (8,9), os autores investigaram essa via e obtiveram que macrófagos mediante KEC possuíam menor presença de RagA em associação a fagolisossomos, assim como redução da fosforilação de S6 e 4E-BP1, moléculas downstream à via mTOR, quando os macrófagos estão em contato com KEC, mas não com LPS. Uma vez reduzida a expressão de RagA nessas células, houve redução da presença de carbonos advindos da KEC na via da síntese de glutationa, demonstrando que o complexo mTORC1 possui um papel regulador nesse processo de reciclagem de nutrientes.
Em relação à distinção entre microrganismos vivos ou não-vivos, verificou-se a expressão de genes associados à síntese de glutationa diante da fagocitose de E. coli morta ou viva (EC), com aumento da expressão desses genes diante da KEC, bem como maior presença de itaconato e produção de espécies reativas de oxigênio (ROS). Por fim, para buscar qual seria o sinal para a diferenciação em relação a vias ativadas pós-fagocitose e, por análise de genes diferencialmente expressos, obteve-se aumento na presença de cAMP após associação entre os macrófagos e KEC, bem como na produção de adenosina por essas células, as quais favorecem a ativação da quinase dependente de AMP (AMPK), representada pela sua maior fosforilação, o que induz redução da ativação do complexo mTORC1, demonstrado pela redução de fosforilação de S6 e 4E-BP1. Essa modulação culmina em respostas antioxidantes, com mais itaconato e menor produção de ROS e IL-1β.
Dessa maneira, Lesbats e colaboradores demonstraram como macrófagos utilizam os nutrientes de microrganismos fagocitados para regular suas funções imunometabólicas e responder ao contexto de infecção.
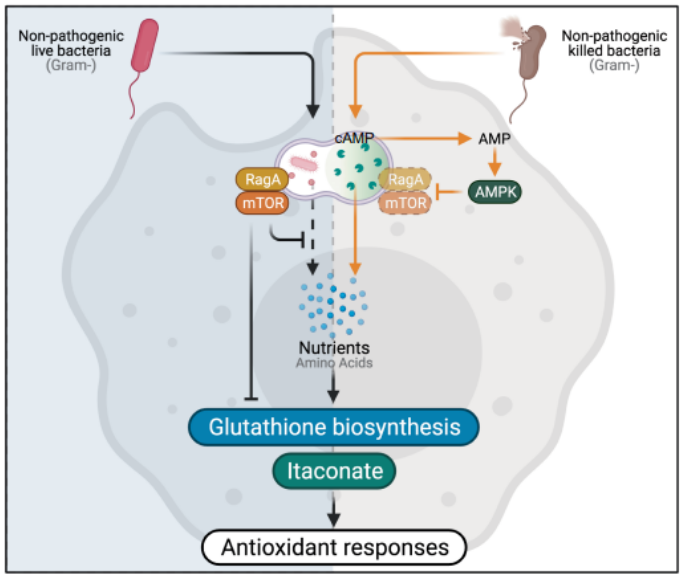
Figura 1. A fagocitose de bactérias ainda vivas induz a ativação do complexo mTORC1 (RagA-mTOR), o que inibe a obtenção de nutrientes advindos da bactéria, implicando na redução da biossíntese de glutationa e itaconato por essas células. Por outro lado, ao fagocitar uma bactéria não-viável, existe aumento na formação de cAMP por advindo desse microrganismo, que induz a ativação da AMPK, inibindo o complexo mTORC1 e favorecendo a síntese de glutationa e itaconato, indutores de respostas antioxidantes por meio de menor produção de ROS e IL-1β.
Sobre a autora:

Larissa Pinto de Andrade é doutoranda do Programa de Pó s-graduação em Imunologia Básica e Aplicada da Faculdade de Medicina de Ribeirão Preto.
Referências
-
Lesbats, J., Brillac, A., Reisz, J.A. et al. Macrophages recycle phagocytosed bacteria to fuel immunometabolic responses. Nature 640, 524–533 (2025). https://doi.org/10.1038/s41586-025-08629-4
-
Palm, W., Thompson, C. Nutrient acquisition strategies of mammalian cells. Nature 546, 234–242 (2017). https://doi.org/10.1038/nature22379
-
Ryan, D. G. & O’Neill, L. A. J. Krebs cycle reborn in macrophage immunometabolism. Annu. Rev. Immunol. 38, 289–313 (2020). https://doi.org/10.1146/annurev-immunol-081619- 104850
-
Underhill, D., Goodridge, H. Information processing during phagocytosis. Nat Rev Immunol 12, 492–502 (2012). https://doi.org/10.1038/nri3244
-
Moretti, J., Soumit Roy, S., Bozec, D. et al. STING Senses Microbial Viability to Orchestrate Stress-Mediated Autophagy of the Endoplasmic Reticulum. Cell 171, 809-823 (2017). https://doi.org/10.1016/j.cell.2017.09.034.
-
Garaude, J., Acín-Pérez, R., Martínez-Cano, S. et al. Mitochondrial respiratory-chain adaptations in macrophages contribute to antibacterial host defense. Nat Immunol 17, 1037–1045 (2016). https://doi.org/10.1038/ni.3509
-
Commisso, C., Davidson, S., Soydaner-Azeloglu, R. et al. Macropinocytosis of protein is an amino acid supply route in Ras-transformed cells. Nature 497, 633–637 (2013). https://doi.org/10.1038/nature12138
-
Efeyan, A., Comb, W. & Sabatini, D. Nutrient-sensing mechanisms and pathways. Nature 517, 302–310 (2015). https://doi.org/10.1038/nature14190
-
Lawrence, R.E., Zoncu, R. The lysosome as a cellular centre for signalling, metabolism and quality control. Nat Cell Biol 21, 133–142 (2019). https://doi.org/10.1038/s41556-018-0244-7
Outros Artigos
01/07/2025 16h56
Disparidades na expectativa de vida revelam fragilidade do Sistema de Saúde Pública Norte-Americano
20/06/2025 16h48
Neurônios nociceptores suprimem a inflamação mediada por neutrófilos Siglec-F+ induzida por macrófagos alveolares para proteger contra fibrose pulmonar
13/06/2025 11h17
Macrófagos reciclam bactérias fagocitadas para abastecer seu próprio metabolismo
09/06/2025 19h27